Torna su
Il centro di biologia molecolare
SARS-CoV-2: caratteristiche del virus
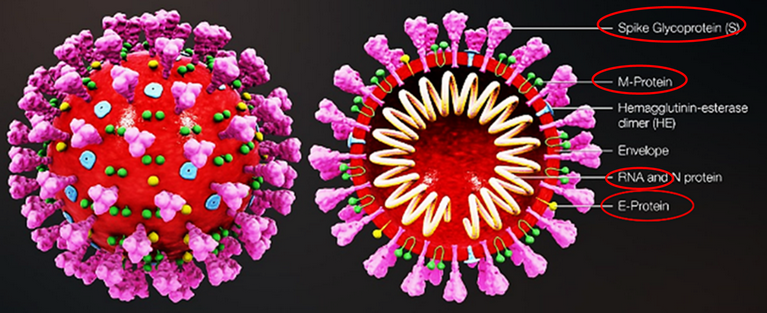
Figura 1. Struttura del SARS-CoV-2. Sono rappresentate le 4 proteine strutturali del virus: la proteina Spike (S), la proteina di membrana (M), la proteina dell’involucro (E) e la proteina del nucleocapside (N). [2]
SARS-CoV-2 nelle acque reflue: un metodo per determinare e monitorare la presenza del virus secondo l’approccio Wastewater-Based Epidemiology
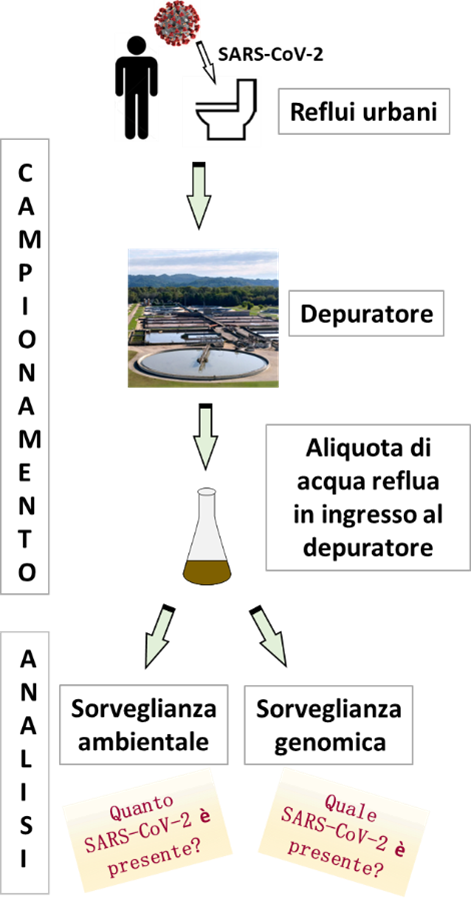
Figura 2. Schema riassuntivo dell’attività di monitoraggio di ARPA Piemonte
SARS-CoV-2: IL MONITORAGGIO DEL VIRUS NELLE ACQUE REFLUE
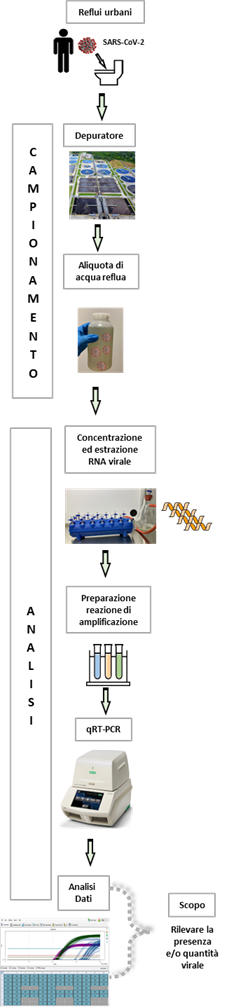
Figura 3. Schema dell’attività di sorveglianza ambientale di SARS-CoV-2: dal campionamento al risultato finale.
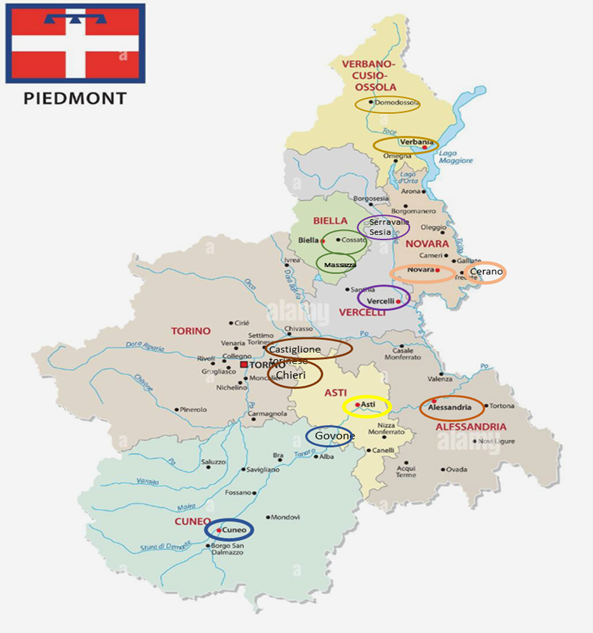
Figura 4 Depuratori coinvolti nel programma di monitoraggio di ARPA Piemonte
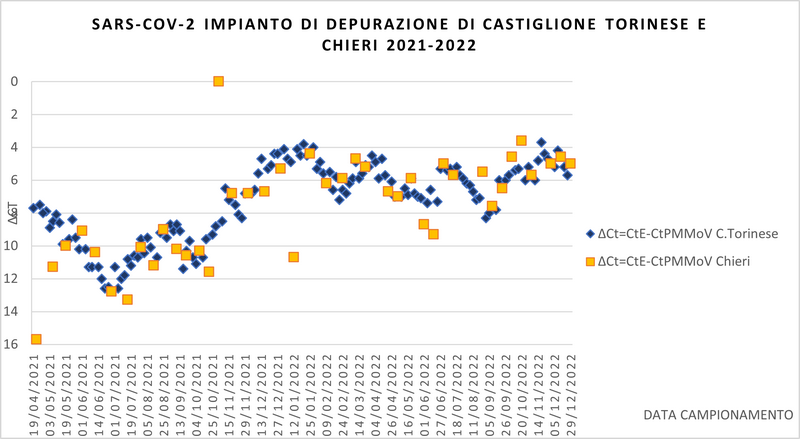
GRAFICO 1: Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Torino.
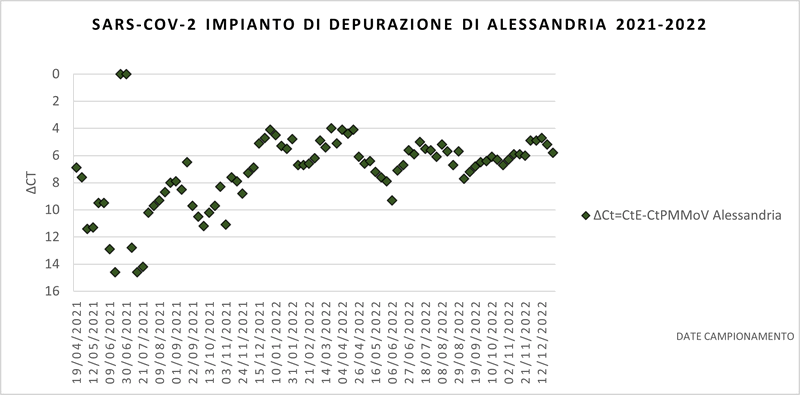

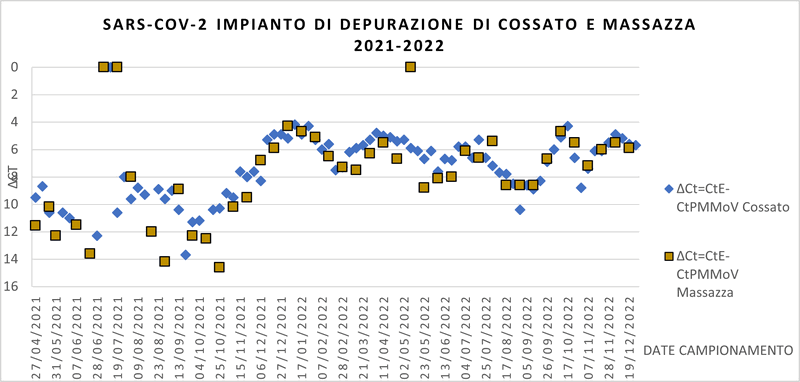
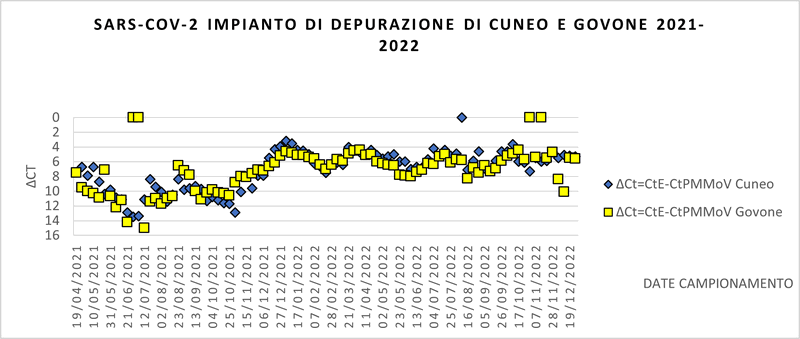


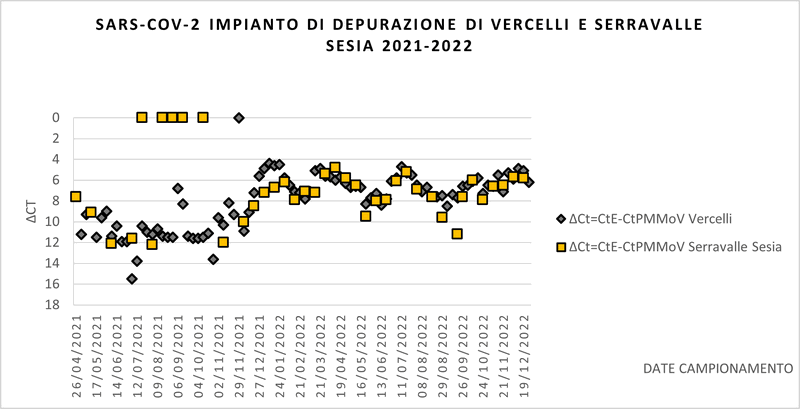
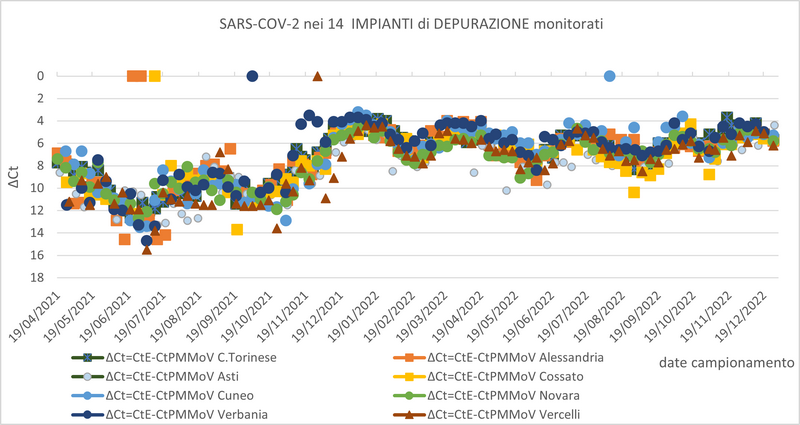

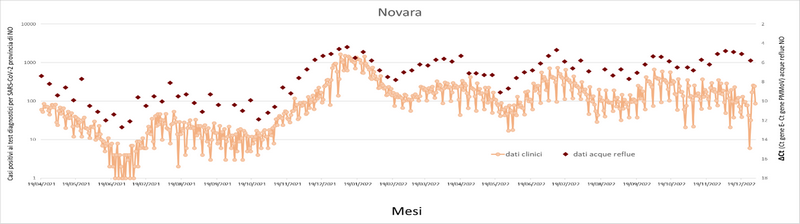
Grafico 11. Confronto tra andamento osservato nelle acque reflue del depuratore di Novara (rombi bordeaux) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Novara (pallini arancioni).
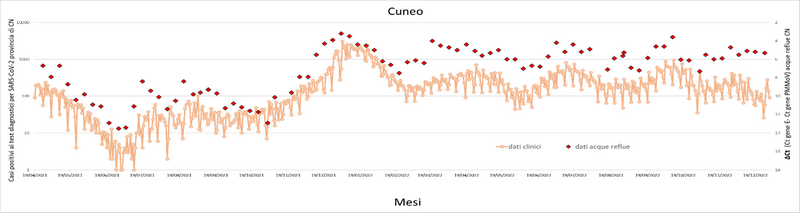
Grafico 12. Confronto tra andamento osservato nelle acque reflue del depuratore di Cuneo (rombi rossi) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Cuneo (pallini arancioni).
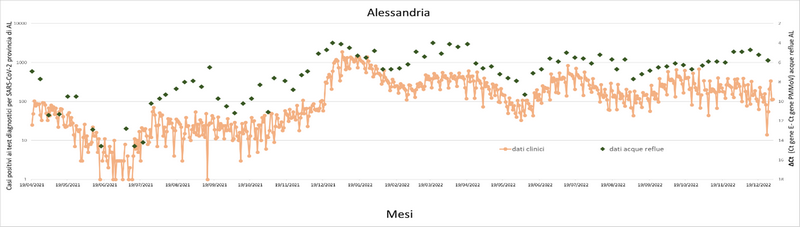
Grafico 13. Confronto tra andamento osservato nelle acque reflue del depuratore di Alessandria (rombi verdi) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Alessandria (pallini arancioni).
SARS-CoV-2: IL MONITORAGGIO GENOMICO DEL VIRUS NELLE ACQUE REFLUE
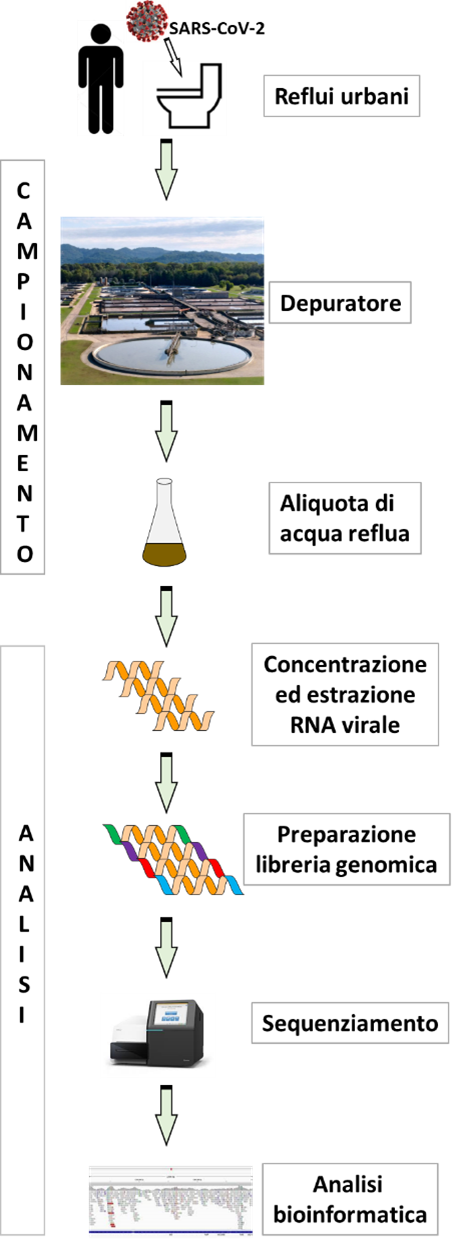
Tabella 2. Descrizione delle diverse categorie di classificazione delle varianti di SARS-CoV-2 [13].
Le varianti classificate come VOC richiedono il massimo livello di attenzione sulla loro diffusione da parte delle autorità sanitarie
Nella tabella 3, viene riportata, come esempio, l’informazione fornita da OMS in merito alla VOC Omicron, classificata come tale nel 2021.

Figura 6. Rappresentazione dei lignaggi e dei clade di SARS-CoV-2.
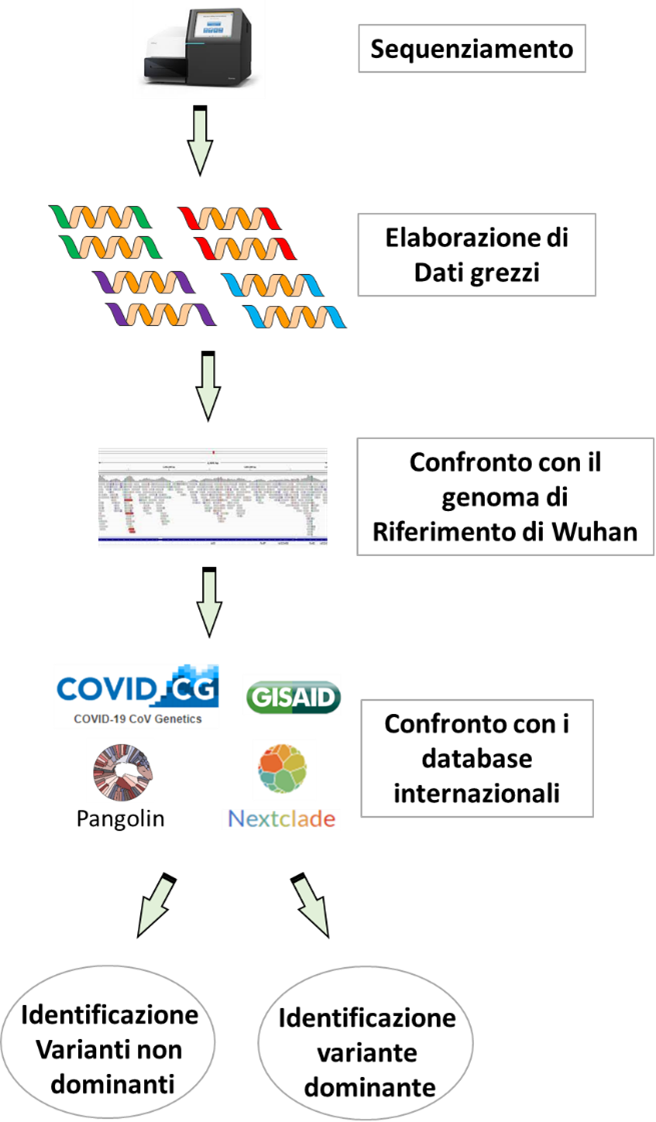
Figura 7. Schematizzazione dell’analisi bioinformatica utilizzata.
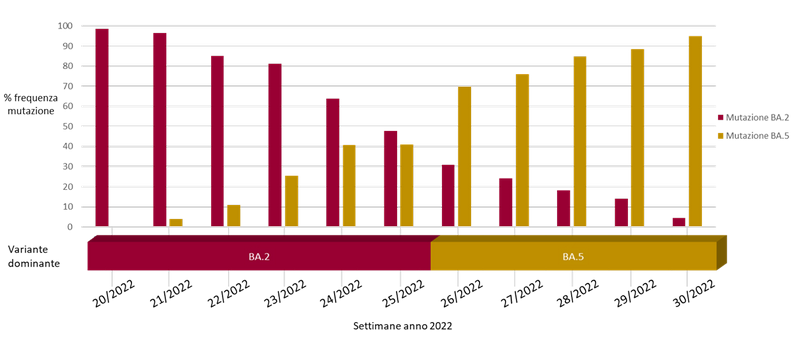
Figura 8. Transizione dalla variante dominante BA.2 alla BA.5 depuratore di Castiglione t.se periodo maggio 2022 – luglio 2022. Il grafico mostra le percentuali di frequenza delle mutazioni distintive della variante BA.2 (color bordeaux) e della variante BA.5 (color ocra) nel periodo compreso tra la settimana 20 e la settimana 30 del 2022.
In figura 9 sono state rappresentate tutte le varianti e le sottovarianti dominanti identificate settimanalmente in Piemonte a partire dalla 49° settimana del 2021, per ognuno dei 4 depuratori. Dal grafico si osserva come per un lungo periodo di tempo, in tutta la regione, si sono susseguite come dominanti le sottovarianti di Omicron BA.1 (da settimana 2-2022 alla settimana 5-2022), BA.2 (dalla settimana 11-2022 alla settimana 24-2022) e BA.5 (da settimana 26-2022 alla settimana 47-2022). La totale dominanza su tutta la regione della variante BA.5 è stata a tratti interrotta dall’emergere delle sottovarianti BA.5.2, BA.2 e BQ.1.

Figura 9. Transizione dalle varianti dominante. Il grafico mostra nel dettaglio l’alternanza delle varianti e sottovarianti dominanti di SARS-CoV-2 identificate nei 4 depuratori piemontesi analizzati a partire dalla settimana 49 del 2021. Per ogni settimana, alla dominanza di ciascun depuratore è stato associato arbitrariamente il valore di ¼, che ha permesso di monitorare la variazione della dominanza complessiva.
CONCLUSIONI
CONTENUTI CORRELATI
BIOLOGIA MOLECOLARE
Il centro di biologia molecolare
Il Centro Regionale di Biologia Molecolare è stato inaugurato il 02/09/2020. La struttura è stata realizzata per dare supporto alle strutture sanitarie per l’analisi di tamponi molecolari per la ricerca di SARS-CoV-2 durante la fase pandemica che persisteva in quel momento. Oltre a questa prima attività, il laboratorio si è attivato fin dalle sue primissime fasi, nella messa a punto di metodi e ricerca di questo virus nelle matrici ambientali come acqua e aria.
Il laboratorio svolge, secondo il Catalogo dei servizi di Arpa Piemonte, le seguenti attività:
B3.10 Monitoraggio pollini
D1.09 Fornitura servizi di prova su acque reflue
D1.24 Fornitura servizi di prova su materiale particellare depositato (particolato atmosferico)
D1.25 Fornitura servizi di prova su aeriformi fissati su supporto solido o liquido
D1.33 Fornitura di servizi di prova su matrici ambientali per la ricerca di legionella
D1.37 Fornitura servizi di prova su tamponi sanitari.
Il personale, che opera in laboratorio, è altamente specializzato in tecniche di indagini biologiche e in particolare in applicazioni di biologia molecolare (come ad es. le analisi in realtime-PCR, sequenziamento genomico), colture cellulari e virali, riconoscimento specie polliniche per citarne alcune.
La dotazione strumentale attualmente presente in laboratorio è per la maggior parte di nuova acquisizione e comprende tutto quanto necessario per applicare molte delle tecniche di indagine molecolare. Alcune delle apparecchiature presenti in laboratorio sono: 2 estrattori automatici di acidi nucleici, 4 Realtime-PCR, un sequenziatore NGS (Illumina MiSeq), un’elettroforesi automatica (Bioanalyzer 2100), un fluorometro (Qubit), un contacellule automatico, diverse tipologie di microscopi (stereomicroscopio, microsopio a fluorescenza, microscopio invertito), 2 centrifughe refrigerate, un’ultracentrifuga (Beckman Optima XE-90), dispensatori di liquidi automatici oltre a 4 cappe biologiche e 2 chimiche. Il grande vantaggio della strumentazione presente in laboratorio è la versatilità e scalabilità che ne permette l’impiego in diverse applicazioni in ambito biologico.
Dopo un primo periodo iniziale in cui sono stati analizzati circa 100.000 tamponi naso-faringei per la ricerca di SARS-CoV-2, il laboratorio ha sviluppato un’attività di ricerca del virus nelle acque reflue (WBE) e su filtri derivanti da campionamenti di aria in differenti ambienti.
Oltre alla ricerca di SARS-CoV-2, sono già iniziate delle sperimentazioni per la ricerca di altri virus e/o patogeni che possono essere ricercati e monitorati nell’ambiente, come ad es. i virus influenzali, con la finalità di sviluppare e testare nuovi approcci epidemiologici utili alla valutazione degli andamenti delle patologie umane.
Il laboratorio svolge, secondo il Catalogo dei servizi di Arpa Piemonte, le seguenti attività:
B3.10 Monitoraggio pollini
D1.09 Fornitura servizi di prova su acque reflue
D1.24 Fornitura servizi di prova su materiale particellare depositato (particolato atmosferico)
D1.25 Fornitura servizi di prova su aeriformi fissati su supporto solido o liquido
D1.33 Fornitura di servizi di prova su matrici ambientali per la ricerca di legionella
D1.37 Fornitura servizi di prova su tamponi sanitari.
Il personale, che opera in laboratorio, è altamente specializzato in tecniche di indagini biologiche e in particolare in applicazioni di biologia molecolare (come ad es. le analisi in realtime-PCR, sequenziamento genomico), colture cellulari e virali, riconoscimento specie polliniche per citarne alcune.
La dotazione strumentale attualmente presente in laboratorio è per la maggior parte di nuova acquisizione e comprende tutto quanto necessario per applicare molte delle tecniche di indagine molecolare. Alcune delle apparecchiature presenti in laboratorio sono: 2 estrattori automatici di acidi nucleici, 4 Realtime-PCR, un sequenziatore NGS (Illumina MiSeq), un’elettroforesi automatica (Bioanalyzer 2100), un fluorometro (Qubit), un contacellule automatico, diverse tipologie di microscopi (stereomicroscopio, microsopio a fluorescenza, microscopio invertito), 2 centrifughe refrigerate, un’ultracentrifuga (Beckman Optima XE-90), dispensatori di liquidi automatici oltre a 4 cappe biologiche e 2 chimiche. Il grande vantaggio della strumentazione presente in laboratorio è la versatilità e scalabilità che ne permette l’impiego in diverse applicazioni in ambito biologico.
Dopo un primo periodo iniziale in cui sono stati analizzati circa 100.000 tamponi naso-faringei per la ricerca di SARS-CoV-2, il laboratorio ha sviluppato un’attività di ricerca del virus nelle acque reflue (WBE) e su filtri derivanti da campionamenti di aria in differenti ambienti.
Oltre alla ricerca di SARS-CoV-2, sono già iniziate delle sperimentazioni per la ricerca di altri virus e/o patogeni che possono essere ricercati e monitorati nell’ambiente, come ad es. i virus influenzali, con la finalità di sviluppare e testare nuovi approcci epidemiologici utili alla valutazione degli andamenti delle patologie umane.
SARS-CoV-2: caratteristiche del virus
SARS-CoV-2 è uno dei membri della famiglia Coronaviridae la quale comprende virus in grado di infettare una vasta gamma di ospiti, tra cui gli esseri umani, producendo una serie di sintomi che possono essere associati a lievi malattie delle vie respiratorie superiori fino a malattie più gravi, anche mortali, come nel caso dei virus SARS e MERS. [1]
Il SARS-CoV-2 è un virus rivestito, con un genoma a singolo filamento di RNA il quale contiene tutte le informazioni necessarie al virus stesso per infettare le cellule ospiti e replicarsi causando la malattia conosciuta come COVID-19.
Nella Figura 1 viene rappresentata in forma schematica la struttura del virus con tutte le sue componenti strutturali e genomiche.
Il SARS-CoV-2 è un virus rivestito, con un genoma a singolo filamento di RNA il quale contiene tutte le informazioni necessarie al virus stesso per infettare le cellule ospiti e replicarsi causando la malattia conosciuta come COVID-19.
Nella Figura 1 viene rappresentata in forma schematica la struttura del virus con tutte le sue componenti strutturali e genomiche.

Figura 1. Struttura del SARS-CoV-2. Sono rappresentate le 4 proteine strutturali del virus: la proteina Spike (S), la proteina di membrana (M), la proteina dell’involucro (E) e la proteina del nucleocapside (N). [2]
La ricerca riporta il fatto che SARS-CoV-2 sia stato ritrovato in campioni di feci sia di pazienti sintomatici che di persone asintomatiche. [3] - [4] - [5]
L’eliminazione del virus anche attraverso le feci fa sì che lo si ritrovi nelle acque reflue in ingresso agli impianti di depurazione e che possano essere quindi eseguite indagini in tale matrice (approccio Wastewater-Based Epidemiology -WBE) [6].
L’eliminazione del virus anche attraverso le feci fa sì che lo si ritrovi nelle acque reflue in ingresso agli impianti di depurazione e che possano essere quindi eseguite indagini in tale matrice (approccio Wastewater-Based Epidemiology -WBE) [6].
SARS-CoV-2 nelle acque reflue: un metodo per determinare e monitorare la presenza del virus secondo l’approccio Wastewater-Based Epidemiology
La Wastewater-Based Epidemiology o epidemiologia basata sulle acque reflue, rappresenta uno strumento epidemiologico utile per monitorare la presenza di sostanze di interesse epidemiologico, quali ad esempio farmaci, sostanze illegali, prodotti del metabolismo umano, nonché agenti responsabili di malattie infettive (poliovirus, enterovirus, etc.). La WBE è dunque potenzialmente in grado di agire come approccio complementare agli attuali sistemi di sorveglianza delle malattie infettive.
L’approccio WBE si è dimostrato uno strumento efficace anche durante la pandemia da SARS-CoV-2 in quanto ha consentito di:
I) Valutare la presenza e/o l’oggettiva quantità del virus in un determinato periodo temporale indipendentemente dai soggetti sottoposti a test (tampone) e dai soggetti asintomatici.
II) prevedere un eventuale picco del contagio;
III) monitorare lo sviluppo di focolai di infezione nelle aree servite da un determinato depuratore.
La Commissione Europea, a seguito dell’inizio della pandemia COVID-19, ha emanato una raccomandazione (Raccomandazione (UE) 2021/472 del 17 marzo 2021) “relativa a un approccio comune per istituire una sorveglianza sistematica del SARS-CoV-2 e delle sue varianti nelle acque reflue nell’UE “ contenente indicazioni per attuare il monitoraggio del SARS-CoV-2 nei reflui urbani, ritenendolo come sopra anticipato un approccio complementare e indipendente rispetto alle altre strategie di sorveglianza sanitaria utilizzate fino a quel momento, in grado di ottenere informazioni provenienti da un ampio bacino di popolazione. [7]
La stessa raccomandazione contiene inoltre indicazioni per attuare il sequenziamento del SARS-CoV-2 presente nei reflui urbani, al fine di monitorare l’insorgere di possibili varianti, la loro diffusione e la loro permanenza nel tempo a livello di popolazione.
Il Centro Regionale di Biologia Molecolare di Arpa Piemonte ha cominciato ad indagare la presenza del virus nelle acque reflue a partire dal 2020 [6] e da aprile 2021 in collaborazione con i Servizi Territoriali di Arpa Piemonte, ha condotto un programma di sorveglianza ambientale sulle acque reflue in ingresso di 14 depuratori Regionali e di sorveglianza genomica sulle acque reflue in ingresso di 4 di questi depuratori (Fig. 2).
L’approccio WBE si è dimostrato uno strumento efficace anche durante la pandemia da SARS-CoV-2 in quanto ha consentito di:
I) Valutare la presenza e/o l’oggettiva quantità del virus in un determinato periodo temporale indipendentemente dai soggetti sottoposti a test (tampone) e dai soggetti asintomatici.
II) prevedere un eventuale picco del contagio;
III) monitorare lo sviluppo di focolai di infezione nelle aree servite da un determinato depuratore.
La Commissione Europea, a seguito dell’inizio della pandemia COVID-19, ha emanato una raccomandazione (Raccomandazione (UE) 2021/472 del 17 marzo 2021) “relativa a un approccio comune per istituire una sorveglianza sistematica del SARS-CoV-2 e delle sue varianti nelle acque reflue nell’UE “ contenente indicazioni per attuare il monitoraggio del SARS-CoV-2 nei reflui urbani, ritenendolo come sopra anticipato un approccio complementare e indipendente rispetto alle altre strategie di sorveglianza sanitaria utilizzate fino a quel momento, in grado di ottenere informazioni provenienti da un ampio bacino di popolazione. [7]
La stessa raccomandazione contiene inoltre indicazioni per attuare il sequenziamento del SARS-CoV-2 presente nei reflui urbani, al fine di monitorare l’insorgere di possibili varianti, la loro diffusione e la loro permanenza nel tempo a livello di popolazione.
Il Centro Regionale di Biologia Molecolare di Arpa Piemonte ha cominciato ad indagare la presenza del virus nelle acque reflue a partire dal 2020 [6] e da aprile 2021 in collaborazione con i Servizi Territoriali di Arpa Piemonte, ha condotto un programma di sorveglianza ambientale sulle acque reflue in ingresso di 14 depuratori Regionali e di sorveglianza genomica sulle acque reflue in ingresso di 4 di questi depuratori (Fig. 2).

Figura 2. Schema riassuntivo dell’attività di monitoraggio di ARPA Piemonte
Le informazioni ottenute con le due tipologie di sorveglianza hanno consentito di avere a disposizione un quadro completo e approfondito della situazione pandemica a livello regionale e seguirne l’evoluzione nel tempo.
SARS-CoV-2: IL MONITORAGGIO DEL VIRUS NELLE ACQUE REFLUE
La ricerca dell’RNA virale nelle acque reflue permette di riscontrare la presenza e la quantità di virus in un preciso momento, in modo indipendente dal manifestarsi dei sintomi nella popolazione e dalla propensione delle persone a rivolgersi al sistema sanitario per sottoporsi a test [6].
Il Centro Regionale di Biologia Molecolare di Arpa Piemonte ha analizzato sistematicamente da aprile 2021 le acque reflue in ingresso ai principali collettori dei depuratori del Piemonte, utilizzando un metodo analitico implementato dal Laboratorio e accreditato nel mese di novembre 2022 da ACCREDIA. In Fig. 3 sono rappresentate schematicamente le fasi del processo di monitoraggio.
Il Centro Regionale di Biologia Molecolare di Arpa Piemonte ha analizzato sistematicamente da aprile 2021 le acque reflue in ingresso ai principali collettori dei depuratori del Piemonte, utilizzando un metodo analitico implementato dal Laboratorio e accreditato nel mese di novembre 2022 da ACCREDIA. In Fig. 3 sono rappresentate schematicamente le fasi del processo di monitoraggio.

Figura 3. Schema dell’attività di sorveglianza ambientale di SARS-CoV-2: dal campionamento al risultato finale.
I campioni
I campioni di acque reflue, medi ponderati nelle 24 ore, sono stati prelevati presso i 14 impianti di depurazione dislocati in tutta la Regione (Fig. 4) con cadenze settimanali o quindicinali (Tab. 1).

Figura 4 Depuratori coinvolti nel programma di monitoraggio di ARPA Piemonte
Tabella 1. Popolazione equivalente e frequenza del campionamento per ciascun depuratore.
| Comune in cui è ubicato l’impianto di depurazione | Popolazione Equivalente (PE) | Frequenza campionamento |
|---|---|---|
| Castiglione Torinese | 3,839,940 | Settimanale |
| Chieri | 60,000 | Quindicinale |
| Alessandria | 110,000 | Settimanale |
| Cuneo | 185,000 | Settimanale |
| Govone | 240,000 | Settimanale |
| Novara | 190,000 | Settimanale |
| Cerano | 122,000 | Quindicinale |
| Verbania | 55,000 | Settimanale |
| Domodossola | 32,000 | Quindicinale |
| Vercelli | 80,000 | Settimanale |
| Serravalle Sesia | 84,000 | Quindicinale |
| Asti | 95,000 | Settimanale |
| Cossato | 520,000 | Settimanale |
| Massazza | 37,000 | Quindicinale |
L’analisi
L’approccio analitico adottato dal Laboratorio prevede la ricerca di RNA virale attraverso le seguenti fasi:
1) concentrazione, estrazione, e purificazione degli acidi nucleici totali (TNA)
2) amplificazione di geni virali targets/bersaglio mediante analisi RT-Real Time PCR. [8]
Per quanto riguarda quest’ultima è stato scelto un sistema multiplex RT-Real Time PCR in grado di rilevare oltre ai geni virali target (gene E/gene N) anche l’RNA di Pepper Mild Mottle Virus (PMMoV). Quest’ultimo è un agente patogeno delle piante, comunemente presente nelle feci e non influenzato da fluttuazioni stagionali, caratteristiche queste che lo rendono idoneo quale target per il controllo di processo analitico e come parametro per eventuale normalizzazione dei dati [7].
La presenza poi di un controllo di amplificazione (Internal Amplification Control - IAC) nel sistema multiplex, consente di monitorare l’eventuale presenza di “inibitori” della reazione di PCR, che possono essere presenti in una matrice complessa quale è l’acqua reflua.
1) concentrazione, estrazione, e purificazione degli acidi nucleici totali (TNA)
2) amplificazione di geni virali targets/bersaglio mediante analisi RT-Real Time PCR. [8]
Per quanto riguarda quest’ultima è stato scelto un sistema multiplex RT-Real Time PCR in grado di rilevare oltre ai geni virali target (gene E/gene N) anche l’RNA di Pepper Mild Mottle Virus (PMMoV). Quest’ultimo è un agente patogeno delle piante, comunemente presente nelle feci e non influenzato da fluttuazioni stagionali, caratteristiche queste che lo rendono idoneo quale target per il controllo di processo analitico e come parametro per eventuale normalizzazione dei dati [7].
La presenza poi di un controllo di amplificazione (Internal Amplification Control - IAC) nel sistema multiplex, consente di monitorare l’eventuale presenza di “inibitori” della reazione di PCR, che possono essere presenti in una matrice complessa quale è l’acqua reflua.
RISULTATI OTTENUTI
Da aprile 2021 a tutto il 2022 sono stati processati dal Centro Regionale di Biologia Molecolare 1067 campioni di acque reflue. Nel dettaglio:
N° campioni analizzati anno 2021: 446
N° campioni analizzati anno 2022: 621
Il 98% circa dei campioni analizzati nel corso dei due anni sono risultati positivi per la presenza di SARS-CoV-2.
L’analisi eseguita con metodica Real Time PCR fornisce, per ogni campione, un valore numerico (Ct) [9] associato alla presenza di ognuno dei geni “bersaglio” ricercati (E/N e PMMoV); tale valore può essere utilizzato per definire un andamento della presenza di SARS-CoV-2 nelle reflue per ogni depuratore.
Il dato è fornito come differenza tra Ct target E e Ct target PMMoV che viene rappresentata come ΔCt.
Nei grafici che seguono sono riportati gli andamenti osservati per i 14 depuratori della Regione Piemonte oggetto di monitoraggio.
N° campioni analizzati anno 2021: 446
N° campioni analizzati anno 2022: 621
Il 98% circa dei campioni analizzati nel corso dei due anni sono risultati positivi per la presenza di SARS-CoV-2.
L’analisi eseguita con metodica Real Time PCR fornisce, per ogni campione, un valore numerico (Ct) [9] associato alla presenza di ognuno dei geni “bersaglio” ricercati (E/N e PMMoV); tale valore può essere utilizzato per definire un andamento della presenza di SARS-CoV-2 nelle reflue per ogni depuratore.
Il dato è fornito come differenza tra Ct target E e Ct target PMMoV che viene rappresentata come ΔCt.
Nei grafici che seguono sono riportati gli andamenti osservati per i 14 depuratori della Regione Piemonte oggetto di monitoraggio.

GRAFICO 1: Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Torino.

GRAFICO 2: Andamento della presenza di Sars-CoV-2 in un depuratore della provincia di Alessandria

GRAFICO 3: Andamento della presenza di Sars-CoV-2 in un depuratore della provincia di Asti

GRAFICO 4: Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Biella

GRAFICO 5: Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Cuneo

GRAFICO 6: Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Novara

GRAFICO 7 Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Verbano-Cusio-Ossola

GRAFICO 8: Andamento della presenza di Sars-CoV-2 in 2 depuratori della provincia di Vercelli

GRAFICO 9: Andamento della presenza di Sars-CoV-2 nei 14 depuratori monitorato della Regione Piemonte
I risultati delle analisi sulle acque reflue ottenuti per alcuni impianti di depurazione sono stati inoltre comparati con l’andamento dei nuovi casi positivi giornalieri segnalati a livello di popolazione della provincia servita dal depuratore [10].
Tale confronto è stato eseguito per i maggiori depuratori regionali: Castiglione Torinese, Alessandria, Cuneo e Novara.
Tale confronto è stato eseguito per i maggiori depuratori regionali: Castiglione Torinese, Alessandria, Cuneo e Novara.

Grafico 10. Confronto tra andamento osservato nelle acque reflue del depuratore di Castiglione Torinese (rombi blu) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Torino (pallini arancioni).

Grafico 11. Confronto tra andamento osservato nelle acque reflue del depuratore di Novara (rombi bordeaux) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Novara (pallini arancioni).

Grafico 12. Confronto tra andamento osservato nelle acque reflue del depuratore di Cuneo (rombi rossi) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Cuneo (pallini arancioni).

Grafico 13. Confronto tra andamento osservato nelle acque reflue del depuratore di Alessandria (rombi verdi) e numero di nuovi casi positivi per SARS-CoV-2 nella provincia di Alessandria (pallini arancioni).
Dai grafici sopra riportati, si può osservare che l’andamento della presenza del virus nelle acque reflue segue l’andamento dei nuovi casi positivi riscontrati a livello di popolazione. Pur evidenziando una tendenza in discesa, a fine 2022 il virus risulta ancora presente nelle acque reflue analizzate di tutti i depuratori regionali.
I risultati ottenuti dal Centro Regionale di Biologia Molecolare di Arpa Piemonte dal 2021 a tutto il 2022 hanno dimostrato la potenzialità e l’importanza di tale monitoraggio. La situazione osservata nelle acque reflue ben rappresenta quanto evidenziato dall’analisi di un grande numero di test diagnostici, dimostrandosi quindi un ottimo strumento di monitoraggio su larga scala.
I risultati ottenuti dal Centro Regionale di Biologia Molecolare di Arpa Piemonte dal 2021 a tutto il 2022 hanno dimostrato la potenzialità e l’importanza di tale monitoraggio. La situazione osservata nelle acque reflue ben rappresenta quanto evidenziato dall’analisi di un grande numero di test diagnostici, dimostrandosi quindi un ottimo strumento di monitoraggio su larga scala.
SARS-CoV-2: IL MONITORAGGIO GENOMICO DEL VIRUS NELLE ACQUE REFLUE
Oltre al dato della presenza e/o quantità di SARS-CoV-2 nelle acque reflue, attraverso il sequenziamento del suo RNAè possibile conoscere quali varianti del virus siano presenti in ogni campione di refluo analizzato. L’analisi di sequenziamento consiste nel “leggere” la sequenza dei genomi virali individuando le mutazioni presenti.
Questa attività fa parte della Sorveglianza genomica ambientale (Fig. 5).
Questa attività fa parte della Sorveglianza genomica ambientale (Fig. 5).

Figura 5. Schema dell’attività di sorveglianza genomica ambientale di SARS-CoV-2: dal campionamento all’individuazione di varianti.
Fin dall’inizio della pandemia di COVID-19 sono emersi, per poi evolversi, diversi lignaggi di SARS-CoV-2 [11], alcuni di questi si sono diffusi velocemente e hanno soppiantato quelli precedenti. Queste “nuove versioni” di SARS-CoV-2 sono chiamate varianti [12]; che, in sostanza, sono versioni differenti del virus derivanti da un insieme di mutazioni del suo genoma.
Tutti i virus, in particolare i virus a RNA come il SARS-CoV-2, quando si replicano nelle cellule che hanno infettato creano milioni di copie di sé stessi e accade di frequente che nella replicazione si generino mutazioni nel loro genoma. Soltanto alcune mutazioni, però, portano a cambiamenti nelle caratteristiche del virus conferendogli un vantaggio in termini di trasmissibilità, evasione del sistema immunitario, virulenza, per citarne alcuni. In base a queste caratteristiche, l’Organizzazione Mondiale della Sanità (OMS) classifica e aggiorna costantemente alcune varianti in 3 categorie: Varianti monitorate (Variants Under Monitoring - VUM), Varianti di interesse (Variants Of Interest - VOI) e Varianti di preoccupazione (Variants Of Concern - VOC) (Tab. 2) [13]
Tutti i virus, in particolare i virus a RNA come il SARS-CoV-2, quando si replicano nelle cellule che hanno infettato creano milioni di copie di sé stessi e accade di frequente che nella replicazione si generino mutazioni nel loro genoma. Soltanto alcune mutazioni, però, portano a cambiamenti nelle caratteristiche del virus conferendogli un vantaggio in termini di trasmissibilità, evasione del sistema immunitario, virulenza, per citarne alcuni. In base a queste caratteristiche, l’Organizzazione Mondiale della Sanità (OMS) classifica e aggiorna costantemente alcune varianti in 3 categorie: Varianti monitorate (Variants Under Monitoring - VUM), Varianti di interesse (Variants Of Interest - VOI) e Varianti di preoccupazione (Variants Of Concern - VOC) (Tab. 2) [13]
| CATEGORIA | DESCRIZIONE |
|---|---|
| VUM | Varianti che esibiscono segnali di crescita a livello epidemiologico,modifiche a livello genomico ed evidenze scientifiche preliminari mal’evidenza è debole o non è ancora stata confermata dall’European Centre forDisease and Control (ECDC). Esse devono essere state causa di un focolaioindividuato all’interno della comunità europea o deve esserci evidenza diun’aumentata trasmissione della variante nel resto del mondo. |
| VOI | Varianti che acquisiscono modifiche genomiche, epidemiologiche oevidenze in vitro che possono implicare un impatto significativo sullatrasmissibilità, severità della malattia e sulla situazione epidemiologica.In ogni caso l’evidenza è ancora preliminare o associata a maggior incertezza. |
| VOC | Varianti le cui mutazioni localizzate nella proteina “Spike” nellaparte del “Receptor Binding Domain” aumentano significativamente la suaaffinità con il recettore hACE2 e si associano ad una più rapida diffusionedel virus nella popolazione umana. |
Tabella 2. Descrizione delle diverse categorie di classificazione delle varianti di SARS-CoV-2 [13].
Le varianti classificate come VOC richiedono il massimo livello di attenzione sulla loro diffusione da parte delle autorità sanitarie
Nella tabella 3, viene riportata, come esempio, l’informazione fornita da OMS in merito alla VOC Omicron, classificata come tale nel 2021.
| WHO label | Pango lineage• | GISAID clade | Nextstrain clade | Additional amino acidchanges monitored° | Earliestdocumented samples | Date of designation |
|---|---|---|---|---|---|---|
| Omicron* | B.1.1.529 | GR/484A | 21K, 21L, 21M, 22A, 22B, 22C, 22D |
+S:R346K +S:L452X +S:F486V |
Multiple countries, Nov-2021 | VUM:24-Nov-2021 VOC:26-Nov-2021 |
L’insieme dei lignaggi forma l’albero filogenetico dell’evoluzione del SARS-CoV-2 (Fig. 6 B) che ha tanti rami quante sono le varianti identificate. La versione riassuntiva dell’albero filogenetico è rappresentata dall’albero dei clade [15] (Fig. 6 A) [16]

Figura 6. Rappresentazione dei lignaggi e dei clade di SARS-CoV-2.
L’evoluzione di SARS-CoV-2 può anche essere causata da eventi di ricombinazione virale che avvengono quando i genomi di due varianti di SARS-CoV-2 che hanno infettato contemporaneamente un individuo si uniscono durante il processo di replicazione. Il risultato è una nuova variante che differisce da entrambi i lignaggi da cui deriva, definiti come parentali, come ad es. il clade 22F (Omicron XBB) (Fig. 6A) [17].
Nel corso della pandemia si è appurato che è possibile trovare il virus nei reflui urbani perché esso, dopo essere entrato nell’organismo umano tramite le vie aeree, viene eliminato anche attraverso le feci. La sua ricerca in tale matrice è stata raccomandata dalla Commissione Europea (RACCOMANDAZIONE UE 2021/472) che ha anche fornito indicazioni in merito al sequenziamento come strumento per monitorare l’insorgere di possibili varianti e osservarne la permanenza nel tempo a livello di popolazione.
Per eseguire il sequenziamento genomico può essere adottata la tecnica del Next Generation Sequencing (NGS) [18] o Sequenziamento di Nuova Generazione che è la tipologia di analisi utilizzata dal Centro Regionale di Biologia Molecolare.Nel dettaglio, l’approccio di NGS scelto dal laboratorio è di tipo Targeted Whole viral genome che consiste nel sequenziamento di tutto il genomavirale. Il passo successivo al sequenziamento consiste in un insieme di analisi computazionali che prende il nome di analisi bioinformatica. Nell’analisi bioinformatica le sequenze generate con il sequenziamento vengono confrontate con la sequenza del genoma di riferimento di SARS-CoV-2 universalmente riconosciuto come “SARS-CoV-2-Wuhan”. Questo confronto permette di individuare variazioni nelle sequenze nucleotidiche generate e, mediante l’uso di banche dati riconosciute e condivise a livello internazionale (ad es. GISAID [19], Pangolin [20], Nextclade [21]), di identificare le varianti presenti. L’analisi bioinformatica descritta e da noi applicata permette l’identificazione della variante dominate [22] e l’identificazione di varianti non dominanti [23] circolanti a bassa frequenza (Fig. 7).
Nel corso della pandemia si è appurato che è possibile trovare il virus nei reflui urbani perché esso, dopo essere entrato nell’organismo umano tramite le vie aeree, viene eliminato anche attraverso le feci. La sua ricerca in tale matrice è stata raccomandata dalla Commissione Europea (RACCOMANDAZIONE UE 2021/472) che ha anche fornito indicazioni in merito al sequenziamento come strumento per monitorare l’insorgere di possibili varianti e osservarne la permanenza nel tempo a livello di popolazione.
Per eseguire il sequenziamento genomico può essere adottata la tecnica del Next Generation Sequencing (NGS) [18] o Sequenziamento di Nuova Generazione che è la tipologia di analisi utilizzata dal Centro Regionale di Biologia Molecolare.Nel dettaglio, l’approccio di NGS scelto dal laboratorio è di tipo Targeted Whole viral genome che consiste nel sequenziamento di tutto il genomavirale. Il passo successivo al sequenziamento consiste in un insieme di analisi computazionali che prende il nome di analisi bioinformatica. Nell’analisi bioinformatica le sequenze generate con il sequenziamento vengono confrontate con la sequenza del genoma di riferimento di SARS-CoV-2 universalmente riconosciuto come “SARS-CoV-2-Wuhan”. Questo confronto permette di individuare variazioni nelle sequenze nucleotidiche generate e, mediante l’uso di banche dati riconosciute e condivise a livello internazionale (ad es. GISAID [19], Pangolin [20], Nextclade [21]), di identificare le varianti presenti. L’analisi bioinformatica descritta e da noi applicata permette l’identificazione della variante dominate [22] e l’identificazione di varianti non dominanti [23] circolanti a bassa frequenza (Fig. 7).

Figura 7. Schematizzazione dell’analisi bioinformatica utilizzata.
Presso il Centro Regionale di Biologia Molecolare dell’Arpa Piemonte, nel periodo tra dicembre 2021 e dicembre 2022 sono stati sottoposti a sequenziamento 223 campioni di acque reflue provenienti dagli impianti di depurazione di Castiglione Torinese, Alessandria, Cuneo e Novara. I campionamenti e le analisi di sequenziamento sono stati eseguiti con cadenza settimanale.
Da un’analisi a posteriori sui dati delle sequenze ritrovate nelle acque reflue del depuratore di Castiglione Torinese nel periodo dell’ondata di Omicron (maggio a luglio 2022), si evince come la sottovariante di Omicron BA.5 abbia soppiantato la sottovariante BA.2 (Fig. 8). La dominanza di BA.5 si è avuta dopo 6 settimane dalla comparsa delle prime mutazioni distintive riscontrate (settimana 21/2022).
Da un’analisi a posteriori sui dati delle sequenze ritrovate nelle acque reflue del depuratore di Castiglione Torinese nel periodo dell’ondata di Omicron (maggio a luglio 2022), si evince come la sottovariante di Omicron BA.5 abbia soppiantato la sottovariante BA.2 (Fig. 8). La dominanza di BA.5 si è avuta dopo 6 settimane dalla comparsa delle prime mutazioni distintive riscontrate (settimana 21/2022).

Figura 8. Transizione dalla variante dominante BA.2 alla BA.5 depuratore di Castiglione t.se periodo maggio 2022 – luglio 2022. Il grafico mostra le percentuali di frequenza delle mutazioni distintive della variante BA.2 (color bordeaux) e della variante BA.5 (color ocra) nel periodo compreso tra la settimana 20 e la settimana 30 del 2022.
In figura 9 sono state rappresentate tutte le varianti e le sottovarianti dominanti identificate settimanalmente in Piemonte a partire dalla 49° settimana del 2021, per ognuno dei 4 depuratori. Dal grafico si osserva come per un lungo periodo di tempo, in tutta la regione, si sono susseguite come dominanti le sottovarianti di Omicron BA.1 (da settimana 2-2022 alla settimana 5-2022), BA.2 (dalla settimana 11-2022 alla settimana 24-2022) e BA.5 (da settimana 26-2022 alla settimana 47-2022). La totale dominanza su tutta la regione della variante BA.5 è stata a tratti interrotta dall’emergere delle sottovarianti BA.5.2, BA.2 e BQ.1.

Figura 9. Transizione dalle varianti dominante. Il grafico mostra nel dettaglio l’alternanza delle varianti e sottovarianti dominanti di SARS-CoV-2 identificate nei 4 depuratori piemontesi analizzati a partire dalla settimana 49 del 2021. Per ogni settimana, alla dominanza di ciascun depuratore è stato associato arbitrariamente il valore di ¼, che ha permesso di monitorare la variazione della dominanza complessiva.
Le sequenze delle varianti dominanti ottenute dal laboratorio sono state inoltre caricate sul database Internazionale GISAID [24] in una sezione dedicata alle acque reflue. La condivisione delle informazioni su database “globali” è un ottimo strumento per rendere disponibili i dati all’intera comunità scientifica.
CONCLUSIONI
La sorveglianza ambientale della presenza di SARS-CoV-2 in concerto con quella genomica, consente di avere una visione puntuale e completa in merito a:
Alla luce dei dati ottenuti con le analisi delle acque reflue è possibile affermare che la diffusione del virus ha interessato tutto il territorio regionale e che Omicron è stata la variante più a lungo presente nell’anno 2022.
La frequenza di campionamento adottata (settimanale) e la scelta di ricercare nuove varianti a bassa frequenza, ha invece consentito di individuare sul nascere nuove potenziali varianti dominanti (es. BA2 nel depuratore di Cuneo nella settimana 08/2022 e BA5 nel depuratore di Alessandria e Novara nella settimana 25/2022) e di seguirne l’evoluzione.
L’analisi dettagliata delle varianti a bassa frequenza e il dato relativo alla variante dominante sono stati oggetto di report settimanali pubblicati sul sito istituzionale e di comunicazioni agli enti preposti.
La presenza a fine 2022 delle varianti BA5, BQ.1 e BQ1.19 sta ad indicare che la circolazione del virus è ancora in atto e che il suo genoma sta ancora mutando (Fig. 9).
- diffusione del virus;
- varianti presenti (dominanti e a bassa frequenza)
- progressione della pandemia.
Alla luce dei dati ottenuti con le analisi delle acque reflue è possibile affermare che la diffusione del virus ha interessato tutto il territorio regionale e che Omicron è stata la variante più a lungo presente nell’anno 2022.
La frequenza di campionamento adottata (settimanale) e la scelta di ricercare nuove varianti a bassa frequenza, ha invece consentito di individuare sul nascere nuove potenziali varianti dominanti (es. BA2 nel depuratore di Cuneo nella settimana 08/2022 e BA5 nel depuratore di Alessandria e Novara nella settimana 25/2022) e di seguirne l’evoluzione.
L’analisi dettagliata delle varianti a bassa frequenza e il dato relativo alla variante dominante sono stati oggetto di report settimanali pubblicati sul sito istituzionale e di comunicazioni agli enti preposti.
La presenza a fine 2022 delle varianti BA5, BQ.1 e BQ1.19 sta ad indicare che la circolazione del virus è ancora in atto e che il suo genoma sta ancora mutando (Fig. 9).
CONTENUTI CORRELATI
Bibliografia
[1] Kuldeep Dhama, Sharun Khan, Ruchi Tiwari, Shubhankar Sircar, Sudipta Bhat, Yashpal Singh Malik, Karam Pal Singh, Wanpen Chaicumpa, D. Katterine Bonilla-Aldana, Alfonso J. Rodriguez-Morales, Coronavirus Disease 2019 –COVID-19 doi.org/10.1128/cmr.00028-20
[3] Aravind Natarajan, Soumaya Zlitni, Erin F Brooks, Summer E Vance, Alex Dahlen, Haley Hedlin, Ryan M Park, Alvin Han, Danica T Schmidtke, Renu Verma, Karen B Jacobson, Julie Parsonnet, Hector F Bonilla, Upinder Singh, Benjamin A Pinsky, Jason R Andrews, Prasanna Jagannathan, Ami S Bhatt, Gastrointestinal symptoms and fecal shedding of SARS-CoV-2 RNA suggest prolonged gastrointestinal infection doi.org/10.1016/j.medj.2022.04.001
[4] Furukawa, N.W., Furukawa, N.W., Brooks, J.T., Sobel, J., 2020. Evidence supporting transmission of severe acute respiratory syndrome coronavirus 2 while presymptomatic or asymptomatic. Emerg. Infect. Dis. 2020 (26), E1–E6. doi. org/10.3201/eid2607.201595, 2020
[5] Tindale, L.C., Stockdale, J.E., Coombe, M., Garlock, E.S., Lau, W.Y.V., Saraswat, M., Zhang, L., Chen, D., Wallinga, J., Colijn, C., 2020. Evidence for transmission of covid-19 prior to symptom onset. Elife 9, 1–34.doi.org/10.7554/ eLife.57149
[6] Angelo Robotto, David Lembo, Paola Quaglino, Enrico Brizio, Denis Polato, Andrea Civra, Jessica Cusato, Giovanni Di Perri,2021. Wastewater-based SARS-CoV-2 environmental monitoring for Piedmont, Italy. Environmental Research. doi.org/10.1016/j.envres.2021.111901
[7] RACCOMANDAZIONE (UE) 2021/472 DELLA COMMISSIONEdel 17 marzo 2021, Gazzetta ufficiale dell’Unione Europea.
[13] www.ecdc.europa.eu/en/covid-19/variants-concern
[14] www.who.int/activities/tracking-SARS-CoV-2-variants
[16] clades.nextstrain.org/
[17] www.cdc.gov/coronavirus/2019-ncov/variants/variant-classifications.html
[19] https://gisaid.org/
[20] https://pangolin.cog-uk.io/
[21] https://clades.nextstrain.org/
Sitografia
[2] https://www.unisr.it/news/2020/3/viaggio-al-centro-del-virus-come-e-fatto-sars-cov-2
[10] https://www.salute.gov.it/portale/nuovocoronavirus/dettaglioContenutiNuovoCoronavirus
Glossario
[8] RT-rt-PCR: (Reverse Transcription realtime Polymerase Chain Reaction), è un metodo che amplifica ciclicamente gli acidi nucleici di interesse (target) e utilizzando sonde fluorescenti specifiche permette di analizzare l’andamento della reazione in tempo reale.
[9] Ct: (ciclo soglia) numero di ciclo del processo di amplificazione RT-rt-PCR in cui l’intensità di fluorescenza supera la soglia impostata.
 [11] Lignaggio: un lignaggio è definito come un gruppo di discendenti strettamente legati e accomunati da un antenato in comune
[11] Lignaggio: un lignaggio è definito come un gruppo di discendenti strettamente legati e accomunati da un antenato in comune
[12] Variante: si definisce variante di SARS-CoV-2 una sequenza che presenta delle mutazioni rispetto al genoma di Wuhan.
[15] Clade: si definisce clade un gruppo costituito da un antenato singolo comune e da tutti i discendenti di quell'antenato. Il clade può essere rappresentato o da un'analisi filogenetica o da un cladogramma.
[18] Next-Generation-Sequencing (NGS): si definisce NGS il sequenziamento degli acidi nucleici di seconda generazione rispetto al sequenziamento Sanger. È un tipo di sequenziamento massivo che permette di sequenziare milioni di frammenti di DNA in parallelo.
[22] Variante dominante: variante presente con maggior frequenza nella popolazione rispetto alle altre varianti.
[23] Variante non dominante: variante presente con frequenza minore nella popolazione rispetto alle altre varianti.
[24] GISAID: il database GISAID (Global Initiative on Sharing Avian Influenza Data) promuove la rapida condivisione a livello mondiale dei dati di tutti i virus influenzali e del SARS-CoV-2. Questi dati includono la sequenza genetica, i relativi dati clinici ed epidemiologici associati ai virus che infettano l’uomo e i dati geografici e specie-specifici associati ai virus, per aiutare i ricercatori a capire come i virus si evolvono e si diffondono durante le epidemie e le pandemie [https://gisaid.org/about-us/mission/]. A dimostrazione dell’importanza che ha il sequenziamento di SARS-CoV-2 trovato nelle acque reflue nel monitoraggio delle varianti virali, è stata creata un’area all’interno del database GISAID per depositare i dati generati da tali sequenze.
[1] Kuldeep Dhama, Sharun Khan, Ruchi Tiwari, Shubhankar Sircar, Sudipta Bhat, Yashpal Singh Malik, Karam Pal Singh, Wanpen Chaicumpa, D. Katterine Bonilla-Aldana, Alfonso J. Rodriguez-Morales, Coronavirus Disease 2019 –COVID-19 doi.org/10.1128/cmr.00028-20
[3] Aravind Natarajan, Soumaya Zlitni, Erin F Brooks, Summer E Vance, Alex Dahlen, Haley Hedlin, Ryan M Park, Alvin Han, Danica T Schmidtke, Renu Verma, Karen B Jacobson, Julie Parsonnet, Hector F Bonilla, Upinder Singh, Benjamin A Pinsky, Jason R Andrews, Prasanna Jagannathan, Ami S Bhatt, Gastrointestinal symptoms and fecal shedding of SARS-CoV-2 RNA suggest prolonged gastrointestinal infection doi.org/10.1016/j.medj.2022.04.001
[4] Furukawa, N.W., Furukawa, N.W., Brooks, J.T., Sobel, J., 2020. Evidence supporting transmission of severe acute respiratory syndrome coronavirus 2 while presymptomatic or asymptomatic. Emerg. Infect. Dis. 2020 (26), E1–E6. doi. org/10.3201/eid2607.201595, 2020
[5] Tindale, L.C., Stockdale, J.E., Coombe, M., Garlock, E.S., Lau, W.Y.V., Saraswat, M., Zhang, L., Chen, D., Wallinga, J., Colijn, C., 2020. Evidence for transmission of covid-19 prior to symptom onset. Elife 9, 1–34.doi.org/10.7554/ eLife.57149
[6] Angelo Robotto, David Lembo, Paola Quaglino, Enrico Brizio, Denis Polato, Andrea Civra, Jessica Cusato, Giovanni Di Perri,2021. Wastewater-based SARS-CoV-2 environmental monitoring for Piedmont, Italy. Environmental Research. doi.org/10.1016/j.envres.2021.111901
[7] RACCOMANDAZIONE (UE) 2021/472 DELLA COMMISSIONEdel 17 marzo 2021, Gazzetta ufficiale dell’Unione Europea.
[13] www.ecdc.europa.eu/en/covid-19/variants-concern
[14] www.who.int/activities/tracking-SARS-CoV-2-variants
[16] clades.nextstrain.org/
[17] www.cdc.gov/coronavirus/2019-ncov/variants/variant-classifications.html
[19] https://gisaid.org/
[20] https://pangolin.cog-uk.io/
[21] https://clades.nextstrain.org/
Sitografia
[2] https://www.unisr.it/news/2020/3/viaggio-al-centro-del-virus-come-e-fatto-sars-cov-2
[10] https://www.salute.gov.it/portale/nuovocoronavirus/dettaglioContenutiNuovoCoronavirus
Glossario
[8] RT-rt-PCR: (Reverse Transcription realtime Polymerase Chain Reaction), è un metodo che amplifica ciclicamente gli acidi nucleici di interesse (target) e utilizzando sonde fluorescenti specifiche permette di analizzare l’andamento della reazione in tempo reale.
[9] Ct: (ciclo soglia) numero di ciclo del processo di amplificazione RT-rt-PCR in cui l’intensità di fluorescenza supera la soglia impostata.

[12] Variante: si definisce variante di SARS-CoV-2 una sequenza che presenta delle mutazioni rispetto al genoma di Wuhan.
[15] Clade: si definisce clade un gruppo costituito da un antenato singolo comune e da tutti i discendenti di quell'antenato. Il clade può essere rappresentato o da un'analisi filogenetica o da un cladogramma.
[18] Next-Generation-Sequencing (NGS): si definisce NGS il sequenziamento degli acidi nucleici di seconda generazione rispetto al sequenziamento Sanger. È un tipo di sequenziamento massivo che permette di sequenziare milioni di frammenti di DNA in parallelo.
[22] Variante dominante: variante presente con maggior frequenza nella popolazione rispetto alle altre varianti.
[23] Variante non dominante: variante presente con frequenza minore nella popolazione rispetto alle altre varianti.
[24] GISAID: il database GISAID (Global Initiative on Sharing Avian Influenza Data) promuove la rapida condivisione a livello mondiale dei dati di tutti i virus influenzali e del SARS-CoV-2. Questi dati includono la sequenza genetica, i relativi dati clinici ed epidemiologici associati ai virus che infettano l’uomo e i dati geografici e specie-specifici associati ai virus, per aiutare i ricercatori a capire come i virus si evolvono e si diffondono durante le epidemie e le pandemie [https://gisaid.org/about-us/mission/]. A dimostrazione dell’importanza che ha il sequenziamento di SARS-CoV-2 trovato nelle acque reflue nel monitoraggio delle varianti virali, è stata creata un’area all’interno del database GISAID per depositare i dati generati da tali sequenze.